Page 95 - Perdih, Andrej, Katja Lakota, Alja Prah. 2020. Strukture bioloških molekul. Univerzitetni učbenik z recenzijo in navodila za vaje. Koper: Založba Univerze na Primorskem.
P. 95
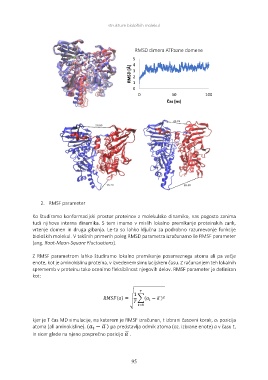
ukture bioloških molekulRMSD [Å]
RMSD dimera ATPazne domene
5
4
3
2
1
0
0 50 100
Čas [ns]
2. RMSF parameter
Ko študiramo konformacijski prostor proteinov z molekulsko dinamiko, nas pogosto zanima
tudi njihova interna dinamika. S tem imamo v mislih lokalno premikanje proteinskih zank,
vrtenje domen in druga gibanja. Le-ta so lahko ključna za podrobno razumevanje funkcije
bioloških molekul. V takšnih primerih poleg RMSD parametra izračunamo še RMSF parameter
(ang. Root-Mean-Square Fluctuations).
Z RMSF parametrom lahko študiramo lokalno premikanje posameznega atoma ali pa večje
enote, kot je aminokislina proteina, v izvedenem simulacijskem času. Z računanjem teh lokalnih
sprememb v proteinu tako ocenimo fleksibilnost njegovih delov. RMSF parameter je definiran
kot:
() = &1 ) − 2 )$
)((
(&*
kjer je T čas MD simulacije, na katerem je RMSF izračunan, t izbrani časovni korak, at pozicija
atoma (ali aminokisline). ( − > ) pa predstavlja odmik atoma (oz. izbrane enote) a v času t,
in sicer glede na njeno povprečno pozicijo $ .
95
RMSD dimera ATPazne domene
5
4
3
2
1
0
0 50 100
Čas [ns]
2. RMSF parameter
Ko študiramo konformacijski prostor proteinov z molekulsko dinamiko, nas pogosto zanima
tudi njihova interna dinamika. S tem imamo v mislih lokalno premikanje proteinskih zank,
vrtenje domen in druga gibanja. Le-ta so lahko ključna za podrobno razumevanje funkcije
bioloških molekul. V takšnih primerih poleg RMSD parametra izračunamo še RMSF parameter
(ang. Root-Mean-Square Fluctuations).
Z RMSF parametrom lahko študiramo lokalno premikanje posameznega atoma ali pa večje
enote, kot je aminokislina proteina, v izvedenem simulacijskem času. Z računanjem teh lokalnih
sprememb v proteinu tako ocenimo fleksibilnost njegovih delov. RMSF parameter je definiran
kot:
() = &1 ) − 2 )$
)((
(&*
kjer je T čas MD simulacije, na katerem je RMSF izračunan, t izbrani časovni korak, at pozicija
atoma (ali aminokisline). ( − > ) pa predstavlja odmik atoma (oz. izbrane enote) a v času t,
in sicer glede na njeno povprečno pozicijo $ .
95